m6A等RNA修饰的主要研究手段是通过MeRIP方法对发生修饰的片段进行富集,其后进行高通量测序分析修饰位点。无论是一篇展示修饰位点分布特点的谱学文章,还是深入研究修饰调控基因表达机制的文章,在高通量测序之后,通常都需要对筛选出来的部分修饰位点进行低通量的MeRIP-qPCR验证。这个实验简单却必不可少,但对于刚刚接触RNA修饰研究的小伙伴来说,可能对MeRIP-qPCR的方法原理、结果含义及展示方法等还存在一些疑问。在已发表文章中又常常是几句话带过,无法解答困惑,回头面对自己手里的数字,还是一头雾水。这次我们就在这里详细谈一谈MeRIP-qPCR的一些相关问题.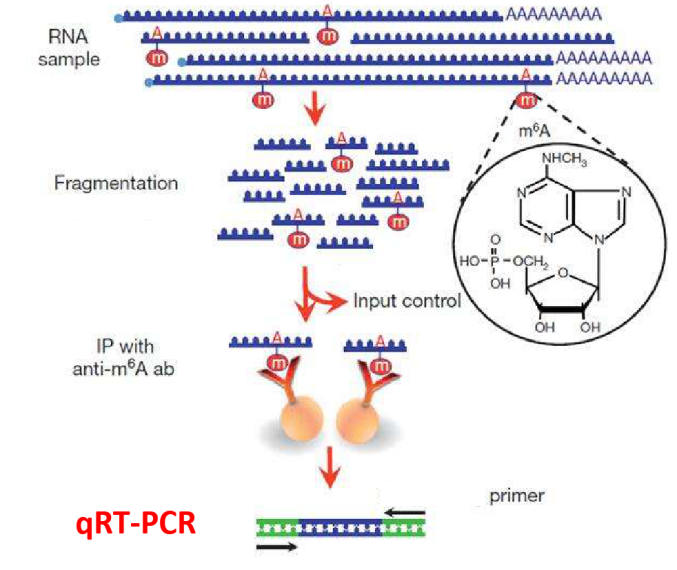
1、MeRIP-qPCR的目的是什么?
MeRIP-qPCR是验证某目标基因上的某个修饰位点(以一小段区域的形式,MeRIP法精确不到单碱基)的修饰水平。得到的结果往往是一个%(IP/Input)的数值,它体现的是该位点的修饰水平(可以理解为:这个转录本表达了很多条,其中这个位点有修饰的有多少条?)。与普通qPCR验证基因表达水平的不同,普通PCR一般是验证基因的表达量(可以理解为:基因转录本表达了多少条?)。
2、MeRIP-qPCR的实验方法?
顾名思义,MeRIP-qPCR就是MeRIP实验后进行的qPCR实验。这里的MeRIP实验同MeRIP-seq测序中MeRIP实验相同,实验基本操作也一致:对RNA进行片段化,然后将片段化后的RNA样品分为二部分:一部分用于免疫共沉淀实验(IP)后作为IP样品,另一部分不进行IP直接作为Input样品。之后再根据实验目的的不同,对两部分RNA片段进行逆转录和qPCR或者建库测序。
3、MeRIP-qPCR的引物如何设计?
(1)从MeRIP-seq的测序结果中筛选获得:
MeRIP-seq报告结果中通常会给出发生甲基化修饰的位置坐标(如云序生物报告中会以chr1:111234-111567的形式给出发生甲基化修饰的区域坐标位置信息)。然后就可以根据位置信息在UCSC genome browser网站(http://genome.ucsc.edu/cgi-bin/hgGateway)获取这段修饰区域的序列(注意基因组版本的选择)。
具体操作如下:
在首页选择对应的物种及基因组版本后,输入修饰区域的位点坐标,点击GO:
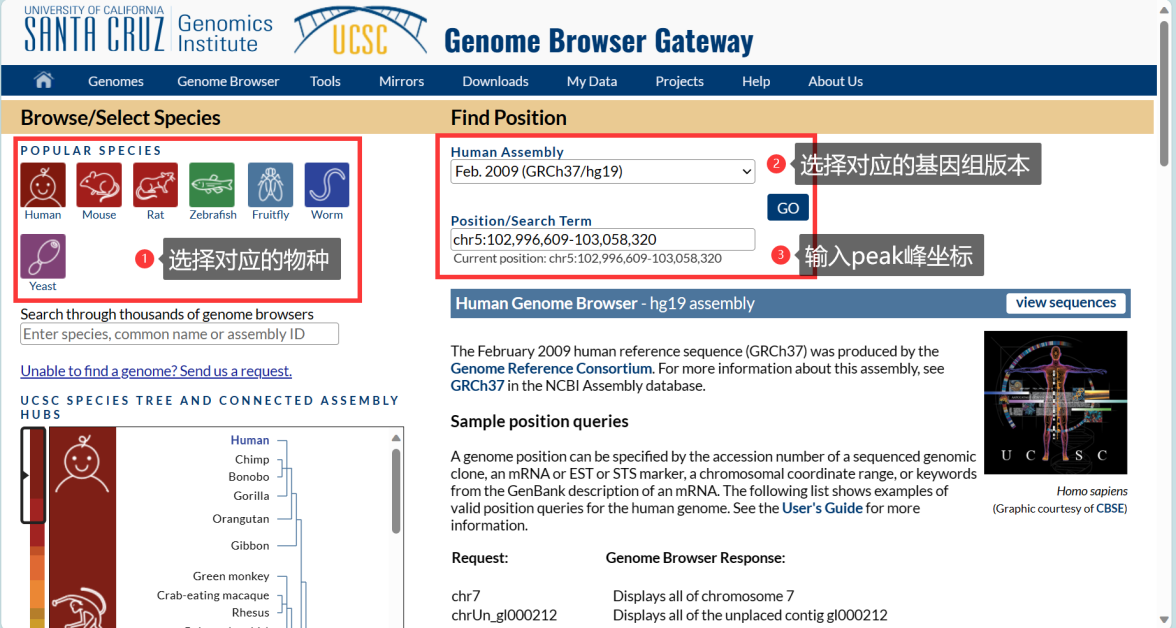
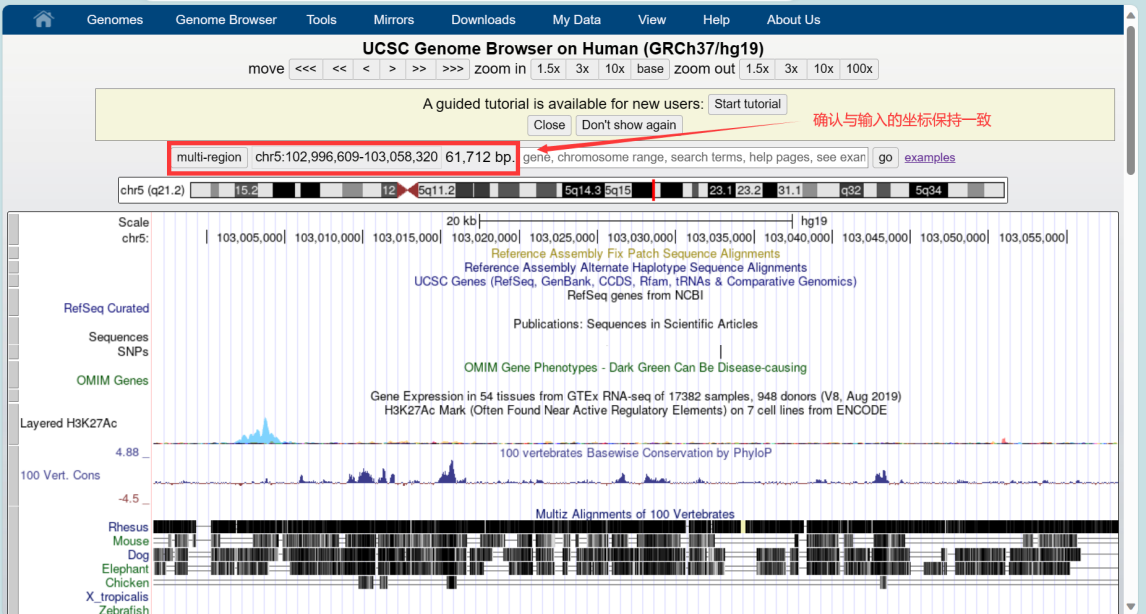
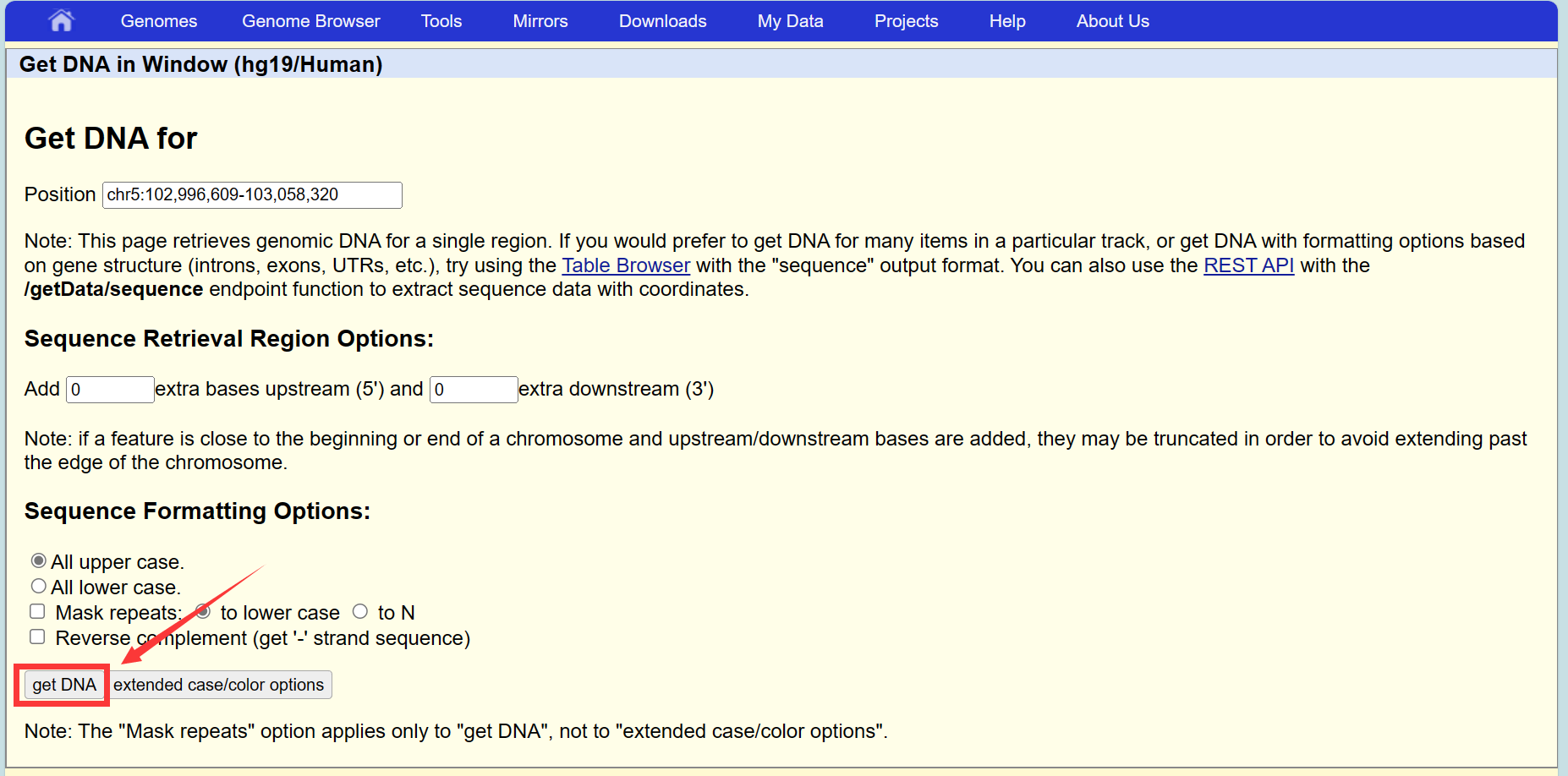
确认位置信息正确后,键盘按V+D,弹出页面,点击get DNA,即可获得修饰区域的序列信息。保存后进行引物的设计:

(2)通过网站预测获得:
如果没有测序结果,想直接通过MeRIP-qPCR验证关注的某基因上是否存在修饰区域,就只能通过预测得到发生修饰概率高的区域位置,再针对位点设计引物,进行qPCR实验。网站的预测通常是基于研究公认的该种修饰具有的motif序列等进行预测,在网站输入目标RNA的序列后,就可生成预测结果。
常用的甲基化预测网站:
m6A RNA甲基化预测网站:http://www.cuilab.cn/sramp/
m5C RNA甲基化预测网站:http://www.rnanut.net/rnam5cfinder/
生成的结果如图:
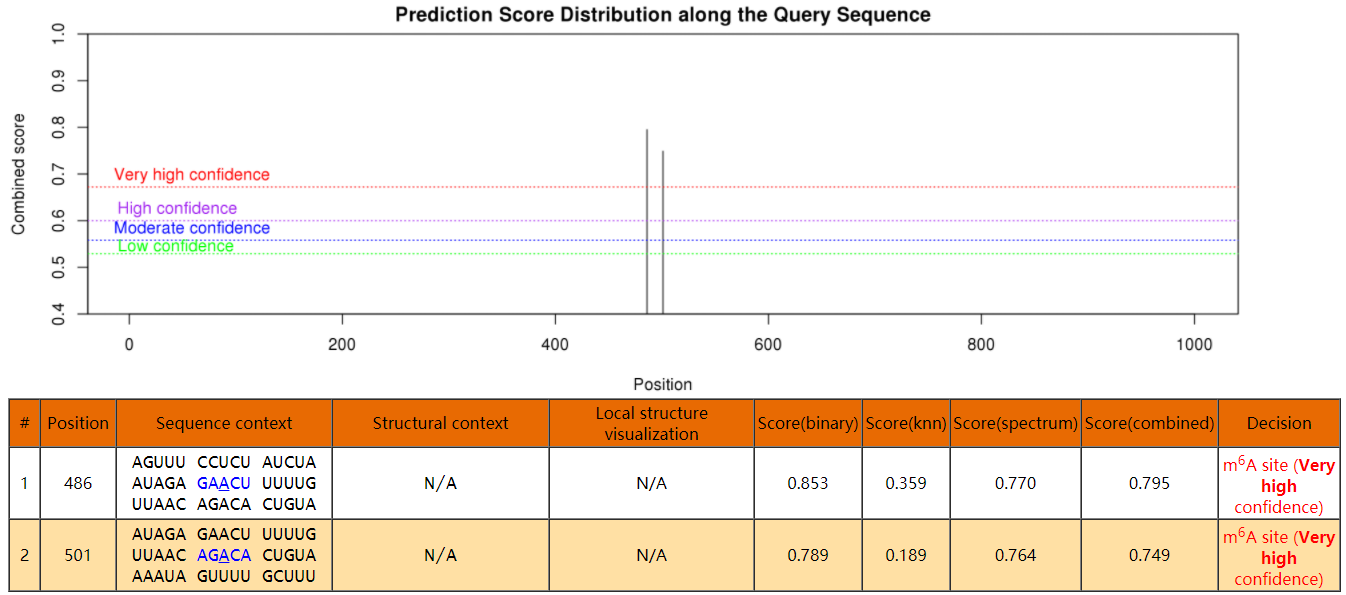
4、MeRIP-qPCR结果的计算
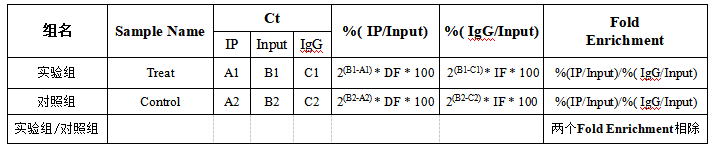
*其中DF和IF是稀释倍数。如最初用于IP和Input的RNA的比例为a:b,则在计算%(IP/Input)值时,需要乘以稀释倍数b/a(经过IP实验得到的RNA量一定少于不做IP实验的Input的RNA量,且逆转录+qPCR的使用的IP和Input的模板量也不同,因此要在计算的时候将差异倍数考虑进去)。一般用于逆转录的Input RNA量为1 μg,则DF=1/(用于IP的RNA的量)。
*P值的计算需要有重复值才能计算。
示例:
只看公式可能不够直观,那就举个例子来详细讲解一下。
例如,实验组和对照组片段化后的总RNA均为21 μg。分别取其中的20 μg的RNA用于IP实验,剩下1 μg的RNA用做Input样品。对IP后的RNA样品及Input RNA进行逆转录(由于IP后RNA样品量较少,建议不测浓度,直接全部用于逆转录)。得到以下Ct值后进行下一步计算:
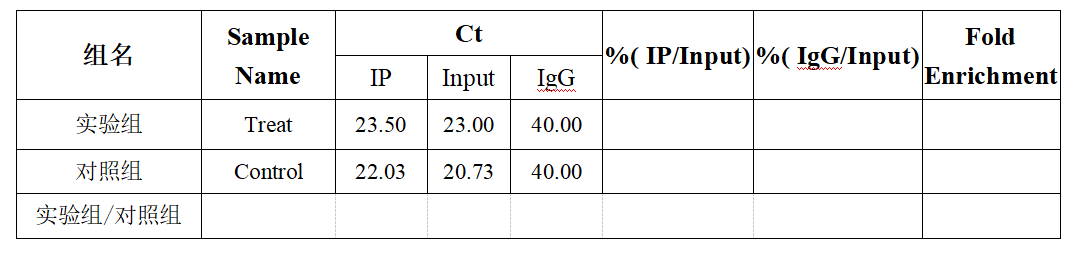
(1)计算稀释倍数:即DF=1/(用于IP的RNA的量)=1/20;
(2)计算%(IP/Input):%(IP/Input)=2(B1-A1)*DF*100
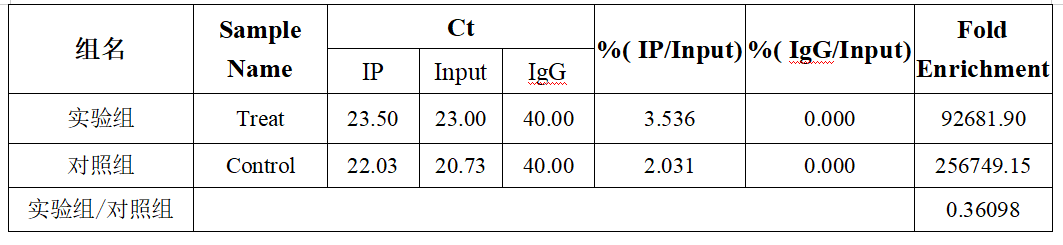
实验组%(IP/Input)=2(23.00-23.50)*(1/20)*100=3.536,%(IgG/Input)=2(23.00-40.00)*(1/20)*100=0.000038547;
对照组%(IP/Input)=2(20.73-22.03)*(1/20)*100=2.031,%(IgG/Input)=2(20.73-40.00)*(1/20)*100=0.000007909;
(在excel中可以使用公式=POWER(2,Input-IP/IgG)*1/20*100进行计算)
(3)计算Fold enrichment:Fold enrichment=%(IP/Input)/%(IgG/Input)
即实验组Fold enrichment=92681.90,对照组Fold enrichment=256749.15;
(4)计算实验组/对照组:实验组Fold enrichment/对照组Fold enrichment=0.36098。
最终计算结果如下表所示:
5、IP/Input/IgG傻傻分不清:我需要做哪些、如何展示?
(1)只做IP和Input:展示%(IP/Input)值
如果有两组样品,展示一个目的基因在两组间的修饰水平。或者一组样品,展示多个基因的修饰水平差异,可以只做IP和Input,展示%(IP/Input)值[1, 2]。
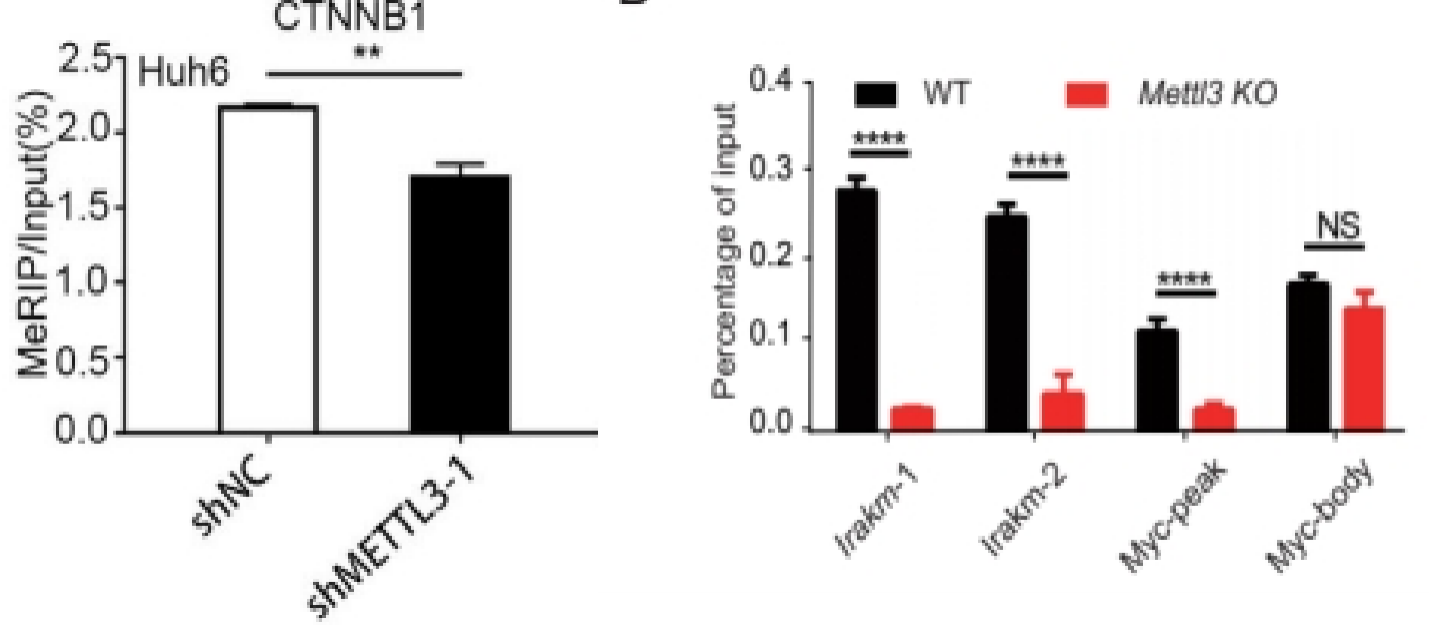
*为了便于直观体现组间的比较,有时会将所有%(IP/Input)数值统一除以某组样品或某个基因的%(IP/Input)数值,使柱状图中该组样品或该基因的柱高变为1,相当于全部以某组样品或某个基因为基准展示相对修饰水平。经这种处理后通常纵坐标会称为relative enrichment、relative level[3, 4]。
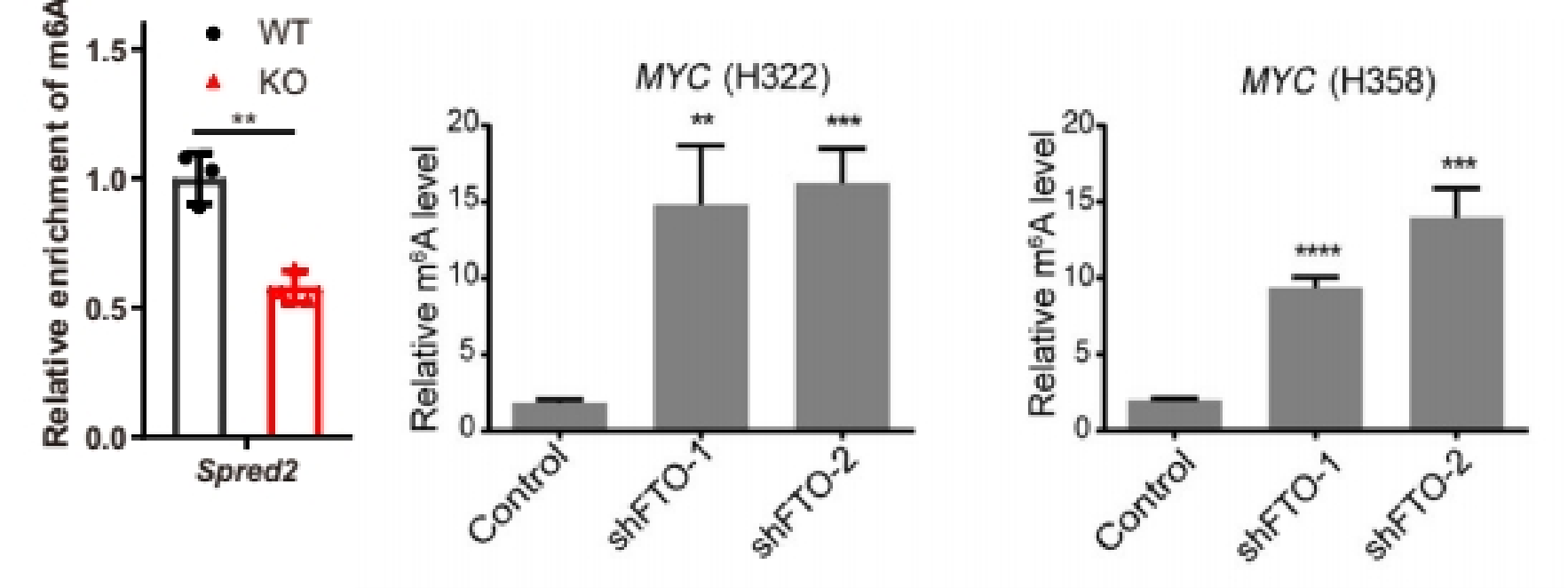
(2)做IP、IgG和Input:展示%(IP/Input)值、%(IgG/Input)值
如果为了严谨的证明实验体系没有问题,或者验证指标只有一个分组/一个基因但需要一个对照,可以同时多做一个IgG,在结果展示%(IP/Input)和%(IgG/Input)两个柱子对照[5, 6]。
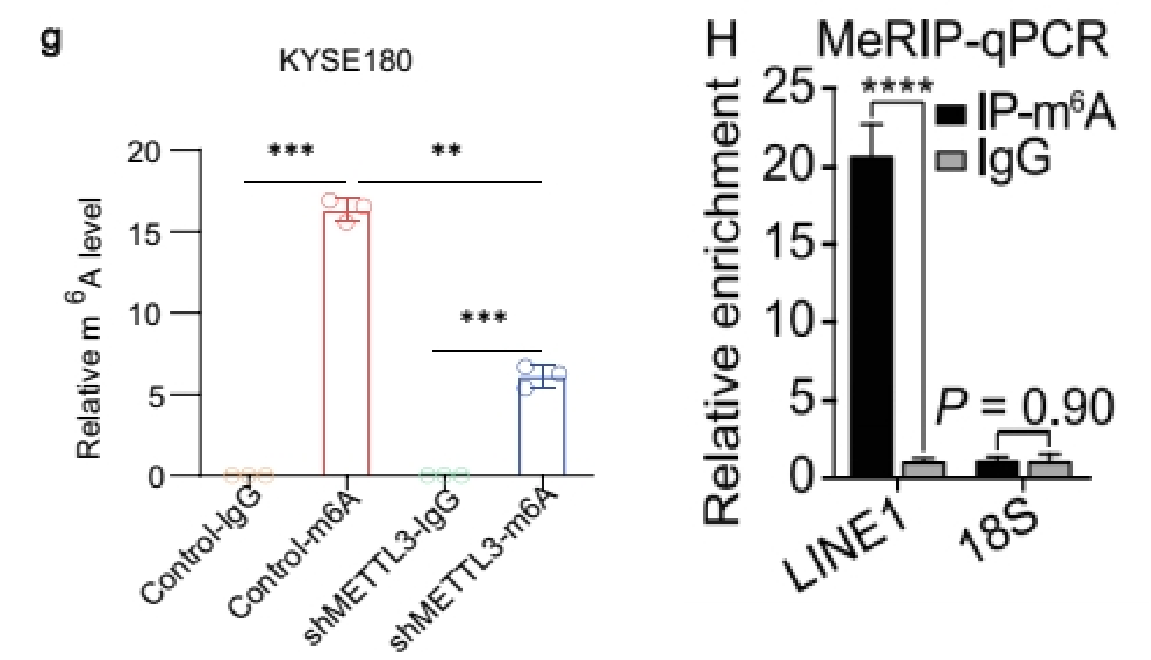
(3)做IP、IgG和Input:展示Fold enrichment值(%(IP/Input)和%(IgG/Input)的比值)
如果做了IgG对照,还可以选择用Fold Enrichment来展示,富集倍数%(IP/Input)/%( IgG/Input),即IP比对照的IgG信号高多少倍,也可以用来体现修饰的水平。通常是分组和基因都是多个时不想一一单独展示IgG[7]。
相信通过上面的学习,你已经基本掌握MeRIP-qPCR这项技能啦!但由于与普通PCR存在很多异同,可能你还是会存在一些疑问。放心!云序生物这就给你解答!
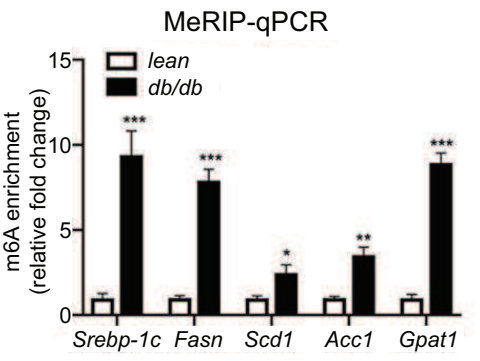
(1)MeRIP-qPCR不需要内参来进行样本间的均一化矫正
传统的RT-qPCR中,每个样本都要做一个内参基因,如哺乳动物会选择GAPDH,而miRNA则会选择U6。那么是不是m6A-IP-qPCR就真的没内参了呢?其实也不是!实际上我们会发现整个input充当了内参基因的概念,一个基因的甲基化水平是无法直接用RIP下来的RNA在做qPCR后直接用Ct值高低来衡量的,需要input中的RNA作为背景。而IgG抗体的加入则是为了排除m6A抗体非特异性结合情况发生。由于有时候m6A抗体不同厂家、保质期、蛋白性能等因素可能会产生非特异性结合的结果,这时候就需要用IgG来排除这部分的干扰。目前市面上使用的绝大部分m6A抗体为兔抗,那么尽量在使用IgG做对照时也使用兔抗来源的IgG。
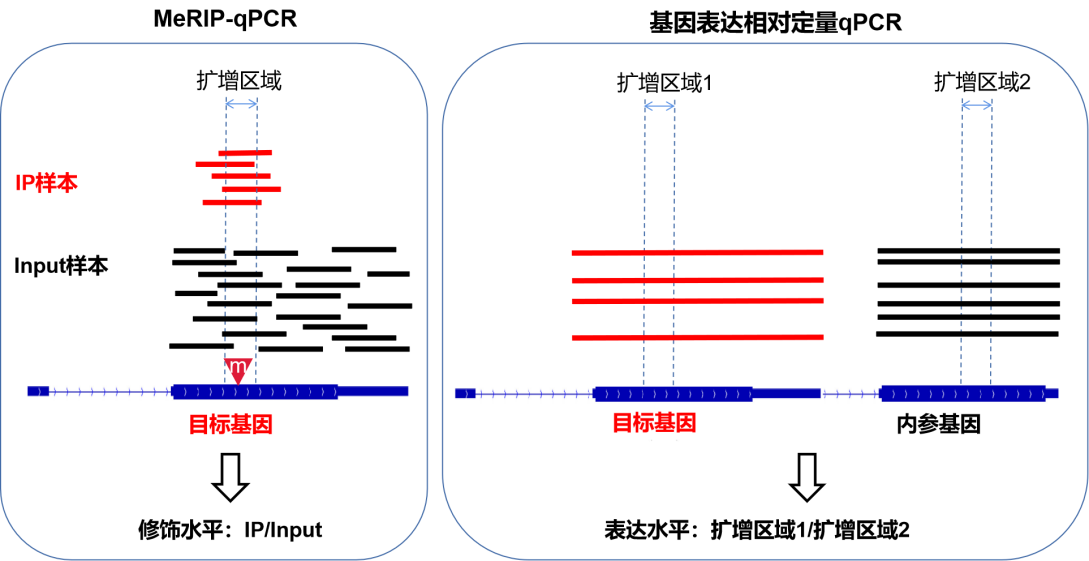
另外,从结果的形式%(IP/Input)也可以看出,MeRIP-qPCR实验侧重看修饰的片段(IP样品qPCR结果)占总片段(Input样品qPCR结果)的比例,实验目的只是要得到这两者的比值就体现修饰水平,因此MeRIP-qPCR不需要内参。
例如:针对目的基因目的位点qPCR,样品A的IP结果0.2,Input结果1;样品B的IP结果0.3,Input结果1.5,得到的%(IP/Input)结果显示两个样品甲基化水平数值都是0.2,修饰水平无差异,到此就达到了目的。
(2)没有公认在不同样品中修饰水平都会稳定的基因可做参照
前面第1点主要从多个样品比较的角度解释了MeRIP-qPCR为何不需要内参基因。而有时也会出现以下情况:1)没有分组对比,只想看下这一个(组)样品中该基因有没有修饰:结果的%(IP/Input)达到多少可算作有修饰?2)想看看MeRIP实验体系有没有问题,想做个已知有修饰的基因当阳性对照看看效果。
针对这两种情况,回答是目前没有一个定值,也没有一个稳定修饰的基因可以做参考。但针对第一种情况,可以在进行实验时,同时做一个非特异性IgG抗体的免疫沉淀富集,对比样品本身的IP和IgG是否有显著差异,放在文章中也可作为该位点有一定程度的修饰的证据。RNA修饰是一个动态可逆的酶促反应,在相同组织细胞中的水平可能都会随着环境千变万化,不同组织中更是差别很大。与普通qPCR检测基因表达时能找到稳定表达的看家基因不同,MeRIP-qPCR没有一个稳定修饰的基因做参照,目前RNA修饰文章也不要求有这种参照。不过也可做IgG对照检测体系的特异性。
因此,在云序生物的GenSeq m6A MeRIP试剂盒中,我们贴心的为客户提供了IgG抗体以及m6A的一对阳参引物和一对阴参引物(根据文献报道:靶向 HEK293 细胞中 m6A 修饰的某段 RNA 区域(Positive Primers)和无 m6A 修饰的某段 RNA 区域(Negative Primers)),供有条件且感兴趣的用户验证实验效率和抗体特异性。但如前面所说,m6A修饰具有很强的细胞类型特异性,无法保证在任何细胞系或样品中,该引物都能作为很好的参照。因此,建议想对实验体系进行进一步自验证的用户,采用HEK293细胞的RNA进行验证实验。

7、MeRIP-qPCR不能看出基因表达量?
那如果在MeRIP-qPCR步骤中直接多扩增一个内参基因,与目的基因Input联合在一起定量表达量是否可行?我们只能说可以参考,不过由于MeRIP实验是有片段化这一步骤的,PCR信号会弱一些,对于验证表达量的目的来说还是推荐采用常规的qPCR步骤会更准确、风险更小。
云序生物服务优势
优势一:云序累计支持客户发表 100+篇RNA修饰SCI论文,合计影响因子 1000+。
优势二:累计完成上万例 RNA甲基化测序样本,全面覆盖医口、农口等各类样本。
优势三:全面检测mRNA和各类非编码RNA(circRNA,lncRNA,Pri-miRNA等)。
优势四:提供m6A一站式服务:m6A RNA修饰整体水平检测、m6A测序、MeRIP-qPCR验证、RIP和RNA pull-down、Ribo-seq、CHIP-seq等技术服务。
优势五:率先研发超微量MeRIP测序技术,RNA量低至500ng起。
优势六:国内较全的RNA修饰测序平台,提供m6A、m5C、m1A、m7G、m3C、O8G、ac4C乙酰化和2'-O-甲基化测序。
云序客户RNA修饰部分文章列表
相关产品
RIP测序
CHIP测序
m6A RNA修饰文章往期回顾
1区,IF=27| 云序m6A MeRIP-seq助力鳞状细胞癌机制研究!
用户文章 1区 lncRNA m6A甲基化测序助力人脂肪干细胞成骨分化的调控机制研究
客户文章|1区,IF=9.995|m6A甲基化测序助力宫颈癌相关HPV病毒研究
用户文章IF=19.568|m5C修饰测序助力NSUN2调控病毒I型干扰素反应的机制研究
Nature 新发现:线粒体 m5C 修饰竟是肿瘤转移的元凶!
用户文章IF=14.9 1区:ac4C乙酰化调节人胚胎干细胞的自我更新
云序用户农口IF20+论文:植物mRNA存在ac4C新型修饰,对RNA稳定性及翻译产生重要影响
用户文章:m7G 甲基化参与调节心肌细胞增殖|新型RNA修饰研究
云序用户植物文章6连发:植物当中各类RNA甲基化测序该怎么发?
庆祝云序用户m1A、m6A、m5C RNA甲基化测序文章三连发
